【matplotlib】プロットを正方形にする方法
いつも忘れるmatplotlibで縦と横の大きさが等しい図の作り方について備忘録的にメモ。
やり方について
簡便的な方法としてplt.figure(figsize=(6, 6))など、figsizeを縦と横で等しくすることで正方形にする方法がある。
簡単な一方で、この方法はタイトルやカラーバー、ラベルなどをつけるなどした場合に必ずしも正方形にならないという欠点がある。
どのようなfigsizeであっても正方形になるようにする方法について以下にメモする。
# モジュールのインポート
import os
import warnings
from urllib import request
import datetime
from sklearn.model_selection import (
KFold,
cross_val_predict,
train_test_split,
check_cv,
)
from sklearn.datasets import load_iris
from sklearn.metrics import r2_score
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.neural_network import MLPRegressor
from sklearn.compose import TransformedTargetRegressor
from sklearn.utils import check_random_state
import optuna
import numpy as np
import pandas as pd
import matplotlib.axes
import matplotlib.pyplot as plt
SEED = 334
データセットのロード
# 配布ページ: https://datachemeng.com/pythonassignment/
url = "https://datachemeng.com/wp-content/uploads/2017/07/logSdataset1290.csv"
dirpath_cache = os.path.abspath("./_cache")
if not os.path.isdir(dirpath_cache):
os.mkdir(dirpath_cache)
fpath_csv_cache = os.path.join(dirpath_cache, os.path.basename(url))
if not os.path.isfile(fpath_csv_cache):
with request.urlopen(url) as response:
content = response.read().decode("utf-8-sig")
with open(fpath_csv_cache, "w", encoding="utf-8-sig") as f:
f.write(content)
df_data = pd.read_csv(fpath_csv_cache, index_col=0)
df_data.head()
| logS | MolWt | HeavyAtomMolWt | ExactMolWt | NumValenceElectrons | NumRadicalElectrons | MaxPartialCharge | MinPartialCharge | MaxAbsPartialCharge | MinAbsPartialCharge | ... | fr_sulfide | fr_sulfonamd | fr_sulfone | fr_term_acetylene | fr_tetrazole | fr_thiazole | fr_thiocyan | fr_thiophene | fr_unbrch_alkane | fr_urea | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CC(N)=O | 1.58 | 59.068 | 54.028 | 59.037114 | 24 | 0 | 0.213790 | -0.369921 | 0.369921 | 0.213790 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| CNN | 1.34 | 46.073 | 40.025 | 46.053098 | 20 | 0 | -0.001725 | -0.271722 | 0.271722 | 0.001725 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| CC(=O)O | 1.22 | 60.052 | 56.020 | 60.021129 | 24 | 0 | 0.299685 | -0.481433 | 0.481433 | 0.299685 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| C1CCNC1 | 1.15 | 71.123 | 62.051 | 71.073499 | 30 | 0 | -0.004845 | -0.316731 | 0.316731 | 0.004845 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| NC(=O)NO | 1.12 | 76.055 | 72.023 | 76.027277 | 30 | 0 | 0.335391 | -0.349891 | 0.349891 | 0.335391 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
5 rows × 197 columns
X = df_data.drop("logS", axis=1)
y = df_data["logS"]
縦と横の範囲が等しい場合
どちらかといえばこちらの方が覚えやすいし、簡単。
オブジェクト指向的な書き方とMATLABっぽい書き方のどちらでも実現できるのが魅力。
縦と横の範囲を手動で固定する必要こそあれ、使用する機会は多い。
たとえば、予実プロットを作成するとき。
予実プロットを作成する
パイプラインについて
最近Pipelineの素晴らしさに気付いたのでちょっと使ってみる
cross validationする場合、本来であれば最初にStandardScalerを適用するのは正しくなくて、各fold内の学習データを使ってスケーリングするのが正しいはずである。
for i_train, i_test in KFold(
n_splits=5, random_state=SEED, shuffle=True
).split(X, y):
# ここにScalingなどをベタ書きすれば可能
pass
上の方法を使えばできることにはできるのだが、面倒だし、コードが長くなり保守性などにも懸念がある。
そこで使えるのがパイプラインということになる。
パイプラインではいくつかの操作をまとめて一つのestimatorのように振る舞うことができる。下記の例でいえばStandardScalerとMLPRegressorをまとめられるということになる。
以下、適当にモデルを作って、Oputnaでハイパーパラメータ探索をしてモデルを構築し、汎化性能の確認のため予実プロット作成する。
X_train, X_test, y_train, y_test = train_test_split(
X, y, random_state=SEED, test_size=0.2
)
pipe = Pipeline(
steps=[
("scaler", StandardScaler()),
(
"mlp",
TransformedTargetRegressor(
regressor=MLPRegressor(), transformer=StandardScaler()
),
),
]
)
pipe
Pipeline(steps=[('scaler', StandardScaler()),
('mlp',
TransformedTargetRegressor(regressor=MLPRegressor(),
transformer=StandardScaler()))])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
Pipeline(steps=[('scaler', StandardScaler()),
('mlp',
TransformedTargetRegressor(regressor=MLPRegressor(),
transformer=StandardScaler()))])StandardScaler()
TransformedTargetRegressor(regressor=MLPRegressor(),
transformer=StandardScaler())MLPRegressor()
MLPRegressor()
StandardScaler()
StandardScaler()
Optunaによるハイパーパラメータ探索
class Objective:
def __init__(
self,
estimator,
X,
y,
cv=None,
random_state=None,
n_jobs=None,
max_layers: int = 10,
):
self.estimator = estimator
self.X = X
self.y = y
self.rng_ = check_random_state(random_state)
self.cv = check_cv(cv)
self.n_jobs = n_jobs
self.max_layers = max_layers
self.fixed_params = {
"mlp__regressor__max_iter": 10000,
"mlp__regressor__shuffle": True,
"mlp__regressor__random_state": self.rng_,
}
def __call__(self, trial: optuna.trial.Trial):
params = {
"n_layers": trial.suggest_int("n_layers", 1, self.max_layers),
"mlp__regressor__activation": trial.suggest_categorical(
"mlp__regressor__activation",
["identity", "logistic", "tanh", "relu"],
),
"mlp__regressor__solver": trial.suggest_categorical(
"mlp__regressor__solver",
["lbfgs", "sgd", "adam"],
),
"mlp__regressor__alpha": trial.suggest_float(
"mlp__regressor__alpha",
1e-5,
1e-1,
log=True,
),
"mlp__regressor__learning_rate": trial.suggest_categorical(
"mlp__regressor__learning_rate",
["constant", "invscaling", "adaptive"],
),
"mlp__regressor__learning_rate_init": trial.suggest_float(
"mlp__regressor__learning_rate_init",
1e-5,
1e-1,
log=True,
),
"mlp__regressor__early_stopping": trial.suggest_categorical(
"mlp__regressor__early_stopping", [True, False]
),
}
for i in range(1, self.max_layers + 1):
params[f"layer{i}_size"] = trial.suggest_int(
f"layer{i}_size", 1, 100
)
# 5-fold cross-validation
y_oof = cross_val_predict(
self.get_estimator(params),
self.X,
self.y,
cv=self.cv,
n_jobs=self.n_jobs,
)
return r2_score(self.y, y_oof)
def get_estimator(self, params: dict):
n_layers = params.pop("n_layers")
layer_sizes = [
params.pop(f"layer{i}_size") for i in range(1, self.max_layers + 1)
]
params["mlp__regressor__hidden_layer_sizes"] = layer_sizes[:n_layers]
self.estimator.set_params(**params, **self.fixed_params)
return self.estimator
def get_best_estimator(
self, study: optuna.study.Study, refit: bool = True
):
params = study.best_params
estimator = self.get_estimator(params)
if refit:
estimator.fit(self.X, self.y)
return estimator
objective = Objective(
pipe,
X_train,
y_train,
cv=KFold(n_splits=5, random_state=SEED, shuffle=True),
n_jobs=-1,
random_state=SEED,
max_layers=10,
)
optuna.logging.disable_default_handler()
t1 = datetime.datetime.now()
# ignore warnings
study = optuna.create_study(
direction="maximize", sampler=optuna.samplers.TPESampler(seed=SEED)
)
study.optimize(objective, n_trials=20, n_jobs=1)
t2 = datetime.datetime.now()
print(f"elapsed time: {t2 - t1}")
optuna.logging.enable_default_handler()
elapsed time: 0:01:42.057029
with warnings.catch_warnings():
warnings.simplefilter(
"ignore", category=optuna.exceptions.ExperimentalWarning
)
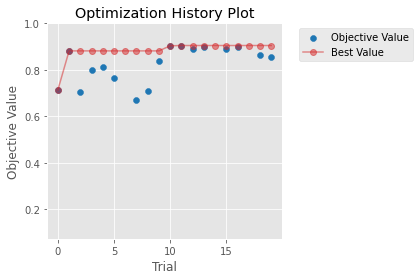
ax: matplotlib.axes.Axes = (
optuna.visualization.matplotlib.plot_optimization_history(study)
)
sr_value = study.trials_dataframe()["value"]
ax.set_ylim(max(sr_value.mean() - sr_value.std() * 2, -1), 1)
print(f"{sr_value.max():.3g}")
ax.figure.tight_layout()
0.905
best_estimator = objective.get_best_estimator(study)
print(study.best_params)
best_estimator.fit(X_train, y_train)
{'n_layers': 1, 'mlp__regressor__activation': 'logistic', 'mlp__regressor__solver': 'adam', 'mlp__regressor__alpha': 0.00418742891311691, 'mlp__regressor__learning_rate': 'constant', 'mlp__regressor__learning_rate_init': 0.000534570171772523, 'mlp__regressor__early_stopping': False, 'layer1_size': 63, 'layer2_size': 91, 'layer3_size': 1, 'layer4_size': 3, 'layer5_size': 98, 'layer6_size': 48, 'layer7_size': 5, 'layer8_size': 4, 'layer9_size': 11, 'layer10_size': 73}
Pipeline(steps=[('scaler', StandardScaler()),
('mlp',
TransformedTargetRegressor(regressor=MLPRegressor(activation='logistic',
alpha=0.00418742891311691,
hidden_layer_sizes=[63],
learning_rate_init=0.000534570171772523,
max_iter=10000,
random_state=RandomState(MT19937) at 0x13F043B40),
transformer=StandardScaler()))])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
Pipeline(steps=[('scaler', StandardScaler()),
('mlp',
TransformedTargetRegressor(regressor=MLPRegressor(activation='logistic',
alpha=0.00418742891311691,
hidden_layer_sizes=[63],
learning_rate_init=0.000534570171772523,
max_iter=10000,
random_state=RandomState(MT19937) at 0x13F043B40),
transformer=StandardScaler()))])StandardScaler()
TransformedTargetRegressor(regressor=MLPRegressor(activation='logistic',
alpha=0.00418742891311691,
hidden_layer_sizes=[63],
learning_rate_init=0.000534570171772523,
max_iter=10000,
random_state=RandomState(MT19937) at 0x13F043B40),
transformer=StandardScaler())MLPRegressor(activation='logistic', alpha=0.00418742891311691,
hidden_layer_sizes=[63], learning_rate_init=0.000534570171772523,
max_iter=10000, random_state=RandomState(MT19937) at 0x13F043B40)MLPRegressor(activation='logistic', alpha=0.00418742891311691,
hidden_layer_sizes=[63], learning_rate_init=0.000534570171772523,
max_iter=10000, random_state=RandomState(MT19937) at 0x13F043B40)StandardScaler()
StandardScaler()
y_pred_on_train = best_estimator.predict(X_train)
y_pred_on_test = best_estimator.predict(X_test)
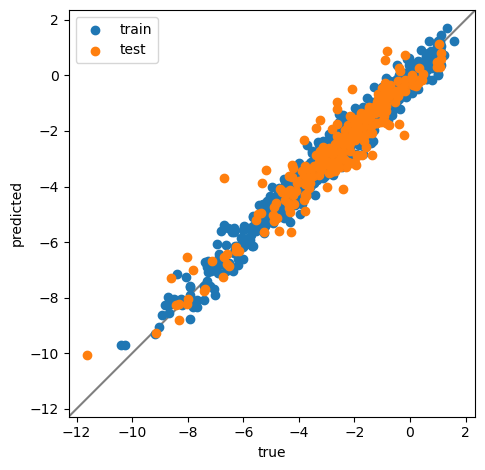
本題の予実プロット
plt.rcParams.update(plt.rcParamsDefault)
fig, ax = plt.subplots()
ax.scatter(y_train, y_pred_on_train, label="train")
ax.scatter(y_test, y_pred_on_test, label="test")
alpha = 0.05 # 余白の割合
range_ = min(
[
np.nanmin(np.array(_y).ravel())
for _y in (y_train, y_test, y_pred_on_train, y_pred_on_test)
]
), max(
[
np.nanmax(np.array(_y).ravel())
for _y in (y_train, y_test, y_pred_on_train, y_pred_on_test)
]
)
offset = (range_[1] - range_[0]) * alpha
plot_range_ = range_[0] - offset, range_[1] + offset
ax.plot(*[plot_range_] * 2, color="gray", zorder=0.5)
ax.set_xlim(*plot_range_)
ax.set_ylim(*plot_range_)
ax.set_xlabel("true")
ax.set_ylabel("predicted")
print(f"R2 on train: {r2_score(y_train, y_pred_on_train):.3g}")
print(f"R2 on test: {r2_score(y_test, y_pred_on_test):.3g}")
# ******* ここが今回の本題 *******
ax.set_aspect("equal", adjustable="box")
# ******* ここが今回の本題 *******
ax.legend()
fig.tight_layout()
R2 on train: 0.97
R2 on test: 0.911
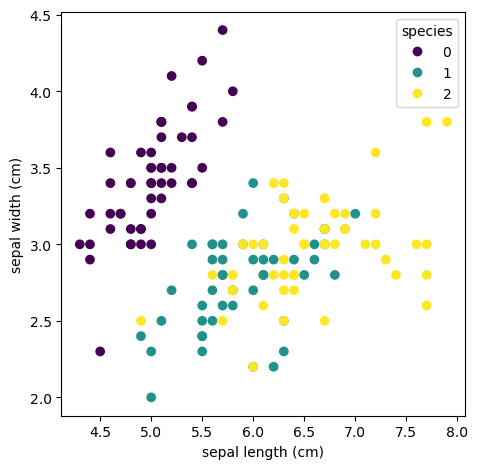
縦と横で違う範囲の場合
前半が脇道に逸れた分長くなってしまったので後半は簡単に。
あやめのデータの分布を確認する場合を例にとって、縦と横で違う範囲の場合にプロットを正方形にする。
iris = load_iris(as_frame=True)
df_iris = iris.data
df_iris.head()
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | |
|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 |
fig, ax = plt.subplots(facecolor="white")
mappable = ax.scatter(
df_iris["sepal length (cm)"],
df_iris["sepal width (cm)"],
c=iris.target,
)
ax.set_xlabel("sepal length (cm)")
ax.set_ylabel("sepal width (cm)")
# ******* ここが今回の本題 *******
ax.set_aspect(1.0 / ax.get_data_ratio(), adjustable="box")
# ******* ここが今回の本題 *******
ax.legend(*mappable.legend_elements(), title="species")
fig.tight_layout()
要点まとめ
長くなってしまったので要点だけをまとめる。以下いずれも、ax: matplotlib.axes.Axesであるとする。
(plt.scatterやplt.plotで書いている人は以下のコードの前にax = plt.gca()を書けば実装できる。)
縦と横で同じ範囲の場合
範囲指定 (ax.set_xlimおよびax.set_ylimにより縦と横を同じ範囲に手動で設定) した上で、
ax.set_aspect("equal", adjustable="box")
縦と横で違う範囲の場合
ax.set_aspect(1.0 / ax.get_data_ratio(), adjustable="box")
コメント
縦と横が同じ範囲の場合はequalと指定するだけなので覚えられるのだが、違う範囲の場合、いつもどうやって比率を与えるべきなのか忘れてしまう。
ax.get_data_ratio()が使われているのをこの用途以外で見たことがないのだが、他にあるのだろうか。





