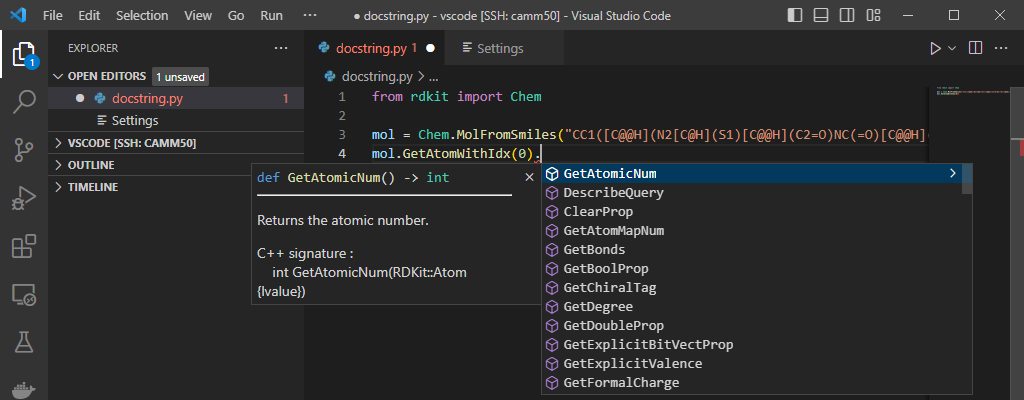
ついにRDKitで型ヒントが出るように!
RDKitは、Pythonで有機分子を取り扱うためにほとんど必須のライブラリであるにもかかわらず、型ヒント機能が脆弱であった。
この度stubが実装されて、とても快適になった。
3行まとめ
rdkit>=2023.09.6でstubが実装された。- これにより型ヒントが強化された。
- さらに、ドキュメントがVSCode上で読めるようにもなった。
これまで
rdkit.Chem.rdchem.Mol (Molオブジェクト) は分子を扱うためのオブジェクトである。Molオブジェクトには、原子の数を数える・芳香族性かどうかを判定する など、その分子の特徴を取り出すたくさんのメソッドが備わっている。
他のライブラリと違って、メソッドがキャメル型で定義されている (のに加えて私が英語に弱い) ため、タイプミスが多く、多くの時間を無駄にしてきた。
Pythonの多くのライブラリでは、型付けがなされていることも多く、正しいメソッド名をレコメンドしてくれるため効率が低下することがない。rdkitにはそれが備わっていなかったため、不便な思いをしていた。 (英語の勉強をしろという話ではあるが…)
今回なにが起こったか
2023.09.6から、自動的にstubを生成する環境が整備された。
stub (スタブ) については私もよくわかっていないのだが、「スタブファイル (.pyiファイル) を準備することで、ソースコード中に型付けされていないライブラリに対して型ヒントを書くことができる」と理解している。
参考: Stub files - mypy 1.10.0 documentation
ARMチップのMacでの注意事項
ARM (Appli silicon) のMacを使用していると、上記のスタブが反映されない。
当方もARMのMacを持っているので確認済み (2023.09.6および2024.03.1-3)。
原因は、rdkit-stubsディレクトリがビルド時になくなってしまっているためらしい; The M chip’s macOS cannot install rdkit-stubs through conda-forge. #7404
応急処置として、pipでインストールするのが良さそう。
基本的にはcondaとpipの混在は避けるべきではある。
当方の環境では、numpyとPillowが入っていればrdkitだけがpipでインストールされたので汚染は最小限と考える。
pip install rdkit
pipでインストールしたrdkit==2024.03.1では正常に補完が機能することを確認した。
コメント
本当に嬉しい。
リンク
やったこと紹介ブログ
プルリク