特別なソフトなしに化合物3Dモデルを動かしてもらう
特別なソフトを使わずに化合物の3次元構造を描画できるようにしたかった。
実際、世の中に化合物を描画するソフトはたくさんあるが、基本的に自分だけでなく見る相手にもそのソフトを使用してもらう必要がある場合がほとんどである。
一般に、会社のPCにソフトをインストールするためには複雑な申請が必要であるケースがある(特に大きな会社)。したがって、そういった会社でも使用できるようなブラウザで化合物の三次元構造をグリグリとマウスで動かしながら触れるような形で出力したいと思った。
どのように実現するか
htmlファイルで書き出せれば良いと気づいた。
これはplotlyでグラフを書き出したときに発想を得た。
まず、ソフトウェアという点でブラウザは標準的に搭載されている可能性の高いソフトウェアのうちの一つである。
加えて、Jupyter Notebook上でマウスで動かせるものは大抵javascriptベースなので実現も容易だと思い検討した。
実際、py3Dmolパッケージは3Dmol.jsをJupyter上で利用するためのwrapperであることからこれをうまいことすればインターネット環境がなくともブラウザだけで3D構造をグリグリ動かせるはずだと考えた。
とりあえずやってみる
import py3Dmol
import rdkit
from rdkit import Chem
from rdkit.Chem import AllChem
環境
for package in (rdkit, py3Dmol):
print(package.__name__, package.__version__)
rdkit 2022.09.1
py3Dmol 1.8.1
SMILESから適当な分子を生成
mol = Chem.MolFromSmiles("BrC(Br)(Br)C(C(C)C)CC=CC(C)(C)C(=O)COc1ccccc1")
mol
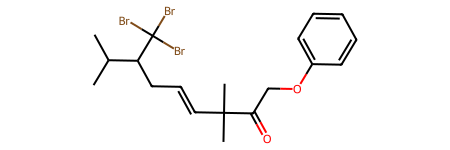
適当に整形
SMILESからmolオブジェクトを生成した段階では2次元かつ水素が付加されていない。
そのため、まず水素を付加し、ある程度reasonableな構造になるようにrdkitに良きに計らってもらう。
水素付加にはChem.AddHs関数、構造をある程度緩和させるにはAllChem.EmbedMolecule関数を用いる。
注意点は前者はコピーを返すが、後者はそのオブジェクト自体を変更すること。
mol = Chem.AddHs(mol)
AllChem.EmbedMolecule(mol)
0
3Dモデルを可視化
jupyter上で確認するには下記のコードで確認できる。
py3dmolでの構造の描画についてはより詳しいサイトがあるので参考されたい。
view = py3Dmol.view(width="100%") # viewオブジェクトの生成
view.addModel(Chem.MolToMolBlock(mol), "sdf", {"keepH": True}) # viewオブジェクトにはMolBlock形式かPDB形式で分子を渡す必要があるため、`Chem.MolToMolBlock`関数でMolBlock形式に変換
view.setStyle({"stick": {"radius":0.25}, "sphere":{"scale":0.35}}) # 分子の表示スタイルを設定
view.show() # viewオブジェクトを表示
生成された分子の3Dモデルはマウスのドラッグで回転し、ホイールで拡大縮小ができる。
ここではviewオブジェクトの生成の過程でwidthを100%と指定したが、これは読み込んだ時のサイトの目一杯まで広げるという意味で、スマホから見てもレイアウトが崩れないように配慮を行った。
もし相手がPCで見るのを想定しているのであればデフォルトの640pxでも問題ないと思う。
ちなみにheightも同様の方法で指定できてデフォルトは480px。
また、分子の表示スタイルで"sphere"方式と"stick"方式を重ね合わせて描画することでより分子とその結合を表せるような気がしたので適当に大きさを手動で合わせてそれっぽくした。
html形式で書き出す
これが多分あまり紹介されていないもの。
viewオブジェクトには内部のための関数として_make_html()がある。これはただ文字列としてhtmlを生成する関数であって保存などはできない。
しかし、この文字列を.htmlとして保存してあげれば良いはず。そう思ってやってみると以下の通り。
with open("py3dmol-example.html", mode="w") as f:
f.write(view._make_html())
実際、上記で生成されたファイルをブラウザソフトで開いてみるとグリグリ動かせるような3Dモデルになっている。
一応確認のためにIPythonメソッドを使ってHTMLとして読み込んでJupyterで表示させてみる。
from IPython.display import HTML
with open("py3dmol-example.html", mode="r") as f:
html = f.read()
HTML(html)
このように動かせるような状態で書き出せていることがわかる。
XYZファイルからやりたいとき
DFT計算の結果の描画など、XYZファイルから描画したいケースは多いのではないかと思う。
カフェインを例にやってみる。
まず、カフェインのxyzファイルの内容を文字列として読み込む。
カフェインのxyzファイルを生成
mol_caffeine = Chem.MolFromSmiles("CN1C(=O)N(C)c2ncn(C)c2C1(=O)") # SMILESからMolオブジェクトを生成
mol_caffeine.SetProp("_Name", "caffeine") # 名前をつける(`_Name`というプロパティを設定する)
mol_caffeine = Chem.AddHs(mol_caffeine) # 水素原子を追加
AllChem.EmbedMolecule(mol_caffeine) # 構造の緩和
Chem.MolToXYZFile(filename="caffeine.xyz", mol=mol_caffeine) # XYZファイルに書き出し
もちろん必ずしも書き出す必要もないが、xyzファイルから読みこむことを想定しているのでわざわざ書き出した。
with open("./caffeine.xyz", mode="r") as f: # 読み込めないときは適切なencodingを指定。
xyz_caffeine = f.read()
描画・書き出しは先ほどと同様。
view = py3Dmol.view(width="100%")
view.addModel(xyz_caffeine, "xyz", {"keepHs": True})
view.setStyle({"stick": {"radius":0.25}, "sphere":{"scale":0.35}}) # 分子の表示スタイルを設定
# htmlファイルで書き出す
with open("caffeine-xyz.html", mode="w") as f:
f.write(view._make_html())
view.show()
二重結合になっていないのがとても気になるが、xyzファイルでは各原子の位置しか保存されていないので仕方がないであろう。
SDファイルでやりたいとき
with Chem.SDWriter("./caffeine.sdf") as writer: # sdfファイルに書き出し
writer.write(mol_caffeine)
先ほどと同様にもちろん必ずしも書き出す必要もないが、SDファイルから読みこむことを想定しているのでわざわざ書き出した。
# 文字列として読み込む
with open("./caffeine.sdf", mode="r") as f:
sdf_caffeine = f.read()
描画・書き出しは先ほどと同様。
view = py3Dmol.view(width="100%")
view.addModel(sdf_caffeine, "sdf", {"keepHs": True})
view.setStyle({"stick": {"radius":0.25}, "sphere":{"scale":0.35}}) # 分子の表示スタイルを設定
# htmlファイルで書き出す
with open("caffeine-sdf.html", mode="w") as f:
f.write(view._make_html())
view.show()
こちらではちゃんと二重結合が描画されている。
コメント
Powerpointにhtmlオブジェクトを添付できるので、送付するパワポファイルに埋め込んであげればダブルクリックでブラウザが起動し、送付先の人に見てもらえる…かもしれない。
うまいこと書けば振動のアニメーションも描画ができるらしいが、自分はやれていない。





